

基于网络调控视角下的慢性阻塞性肺疾病研究
(1. 海南大学数学与统计学院, 海口 570228
2. 海南大学国际旅游与公共管理学院, 海口 570228
3. 海南大学海南省工程建模与统计计算重点实验室, 海口 570228)
摘要: 慢性阻塞性肺疾病COPD作为一种常见且严重危害健康的呼吸系统疾病, 深入解析其发病机制及调控网络意义深远。本文以精准揭示 COPD的调控网络为核心目标, 系统且严谨地探究其与多发病生物途径错综复杂的内在联系。研究紧密结合大量实际病例数据, 并广泛参考前沿权威文献, 运用先进的数据挖掘模型以及精细的网络调控模型, 着力构建蛋白质-蛋白质相互作用PPI网络。在此过程中, 充分利用吸烟暴露体所涵盖的数据信息, 精准识别与之关联的RNA和miRNA, 为后续研究夯实基础。通过综合运用前沿的网络调控与追踪技术, 成功构建起以 latent TGF-β、Wnt、typeI (ACVR1) 、ADRB2、SMAD7和ROCK1为关键节点的COPD调控网络。随后, 采用高精准度的基因表达数据分析手段, 针对这六个与COPD紧密相关的基因展开敏感度评估, 并依据严格的标准将其敏感度从高到低进行排序, 结果表明latent TGF-β敏感度位居首位, 确认为敏感基因。最终, 凭借科学设定各个基因表达的阈值以及制定与之适配的干预方案, 切实达成对COPD病情的有效预警分析, 为该疾病的早期诊断提供关键指向标, 也为后续精准治疗筑牢坚实的科学依据根基, 有望推动COPD临床诊疗水平迈向新高度。
关键词: COPD, 敏感基因, 预警分析, 网络调控, 数据挖掘
DOI: 10.48014/jcss.20241212002
引用格式: 陈秋宇, 李欣蕾, 王怀星, 等. 基于网络调控视角下的慢性阻塞性肺疾病研究[J]. 中国统计科学学报, 2025, 3(1): 1-14.
文章类型: 研究性论文
收稿日期: 2024-12-12
接收日期: 2025-01-12
出版日期: 2025-03-28
1 引言
慢性阻塞性肺疾病(COPD)患者常常面临多重疾病共存的挑战,包括心血管疾病(CVD)、肌肉萎缩、骨质疏松、代谢综合征、抑郁症以及癌症等,这些共病状态显著恶化了患者的健康状况,并可能导致病情的进一步恶化和生命预后的缩短。尽管COPD及其伴随疾病的具体发病机制尚未完全阐明,但已知一些共同的风险因素(如吸烟、缺乏运动)和COPD特有的病理机制(如全身性炎症、组织缺氧)可能涉及影响该病的基因、蛋白质和信号通路,这些因素在特定患者群体中往往有共同出现的倾向。
Ezzie ME等从公共数据库中搜集了与COPD相关的基因、蛋白质、蛋白质间相互作用以及生物标志物等数据,并对此进行一系列的数据挖掘分析[1]。利用这些数据,构建了COPD的疾病网络,其中网络节点代表基因或蛋白质,而边则代表它们之间的相互作用。特别地,微小核糖核酸(miRNAs)作为一类小分子非编码RNA,它们在调节特定基因和蛋白质水平方面发挥着重要作用,与COPD相关的生物途径密切相关。通过对非COPD吸烟者和COPD吸烟者的肺组织进行miRNA和mRNA表达分析。因此,分析各调控网络的敏感度,从而揭示COPD的调控网络以及吸烟对这一网络的影响,同时识别在COPD发病机制中起关键作用的基因或基因组合,这些基因可能成为未来治疗COPD的潜在靶点[2]。
本文利用Reactome数据库对常见蛋白质集进行功能富集分析[7],尤其是针对六个敏感基因之间关系的探讨,进一步探索COPD及其共病中共享基因和蛋白质的功能,并通过为这些敏感基因设定阈值来实现对COPD及其并发症的预警分析。该模型为COPD及其共病的早期诊断、预后评估和治疗提供了新的见解。以敏感基因latent TGF-β为例,对其进行实时监督分析后,设定0.7886的fold change数作为阈值,如果患者的latent TGF-β基因表达的fold change数超过正常人的该值,则被视为超出预警范围;如果低于该阈值,则认为没有超出预警。一旦检测到超出预警的情况,将立即采取相应的干预措施,以此实现对于COPD的早期预警。
2 COPD调控网络推断
通过深入挖掘公共数据库中与慢性阻塞性肺疾病(COPD)相关的基因、蛋白质、蛋白质间的相互作用以及生物标志物等关键数据,在构建COPD的疾病网络后,进一步对未患COPD的吸烟者和患有COPD的吸烟者的肺组织进行mRNA和miRNA的表达检验。对比COPD患者和健康吸烟者的肺组织样本,发现70个miRNA和2667个mRNA在两组之间存在差异表达,这些差异表达的miRNA和mRNA丰富了可能与COPD发病机制相关的生物途径,包括转化生长因子β、Wnt和粘着斑途径。特别是miR-223和miR-1274a在COPD患者中受影响最大,而miR-15b在COPD的GOLD类别中差异表达,并且定位于肺气肿和纤维化两个区域。
为了探究miRNA在COPD患者肺部是否存在差异表达,并进一步验证miRNA表达与mRNA表达之间的相关性,以及它们与COPD发病机制的潜在联系,本研究收集了35份组织样本,并详细记录了受试者的临床信息。这些信息包括年龄、性别、身高、体重和吸烟史,以及肺功能测试结果、病理诊断和圣乔治呼吸问卷(SGRQ)健康调查结果。这些详尽的数据被用于后续的miRNA和mRNA表达分析,具体信息见下表1。
选取用miRNA微阵列和信使RNA微阵列分析技术,以深入探究COPD患者肺部的miRNA和mRNA表达差异。通过使用LIMMA和QVALUE这两个R资源库,对吸烟者和COPD患者样本之间的差异表达进行了严格的统计测试。在分析过程中,特别关注了差异表达的miRNAs和mRNAs,设定了pFDR<0.05作为统计显著性的阈值,并且要求组间表达变化至少达到1.5倍(log2 fold change),这一标准有助于筛选出在COPD中可能发挥重要作用的基因和miRNA,能够更准确地识别出与COPD病理过程相关的分子标记。
表1 研究患者的人口统计学特征[1]
Table 1 Study patients demographics[1]
|
|
正常吸烟者(N[9]) |
黄金1(N[9]) |
黄金2(N[9]) |
黄金4(N[9]) |
P值 |
|
男性(%) |
44 |
42.8 |
44 |
60 |
0.880 |
|
年龄 |
63.22611.43 |
69.7167.39 |
69.1167.30 |
50.9065.62 |
<0.001 |
|
包-年吸烟 |
25.67616.70 |
41.86623.26 |
44.00620.15 |
56.40636.42 |
0.106 |
|
SGRQ分数 |
11.86617.27 |
31.90619.35 |
36.79616.79 |
61.05615.62 |
<0.001 |
|
身体质量指数 |
28.7666.81 |
26.9764.10 |
26.5065.23 |
24.8862.59 |
0.409 |
|
FEV1 PD |
104.22615.50 |
89.00655.77 |
70.5665.05 |
21.2067.96 |
<0.001 |
为了验证miRNA微阵列分析中发现的差异表达的miRNAs,研究采用定量逆转录聚合酶链反应(qRT-PCR)技术。这一验证过程是在相同的RNA样本集上进行的,包括9名正常吸烟者和26名COPD患者。针对个体miRNA进行了qRT-PCR验证,具体包括(A)miR-15b,(B)miR-223,(C)miR-1274a,以及(D)miR-424。这些qRT-PCR实验在两个独立实验中重复进行,以确保结果的可靠性。通过Students t测试,发现与正常吸烟者相比,COPD患者中miR-15b、miR-223、miR-1274a和miR-424的表达存在统计学上的显著差异,这些结果在图1中得到了直观展示,其中p值是用来决定是否拒绝零假设的关键统计量。miR-223和miR-1274a因其在COPD样本中表达量的显著增加,以及miR-15b和miR-424基于靶点预测结果和它们在包括TGF-β信号通路在内的潜在作用,被选为进一步验证的对象。
Ezzie ME等人通过对所有COPD样本(n=26)和所有无气流受限的吸烟者组织样本(n=9)进行qRT-PCR验证,确认了miR-223、miR-1274a、miR-15b和miR-424的表达差异具有统计学意义[1]。这些发现进一步巩固了miRNA微阵列分析的结果,并为这些miRNAs在COPD病理过程中的作用提供了强有力的实验证据。
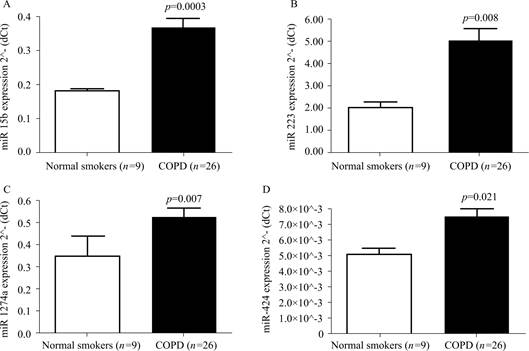
图1 qRT-PCR验证差异表达的microRNAs[1]
Fig.1 qRT-PCR verification of differentially expressed microRNAs[1]
Ezzie ME等人同样也探讨mRNAs在COPD中的差异表达情况[1]。研究特别关注了(A)白细胞介素6(IL-6)、(B)端粒酶相关蛋白1(TEP1)、(C)过氧化氢酶(CAT)以及(D)母亲对抗脱(果蝇,SMAD7)四个基因。通过qRT-PCR技术,对这些基因的表达水平进行了验证,结果如图2所示。接着选择对IL-6、TEP1、CAT和SMAD7进行qRT-PCR进行验证,因为它们在COPD的发病机制中扮演着重要角色。尤其是SMAD7,已知其在COPD的发病机制中被激活,微阵列分析显示其表达显著降低,这一点通过qRT-PCR得到了验证;与无气流限制的吸烟者相比,COPD样本中IL-6的表达增加,而CAT的表达减少;作为一种与端粒酶活性相关的哺乳动物蛋白,TEP1在COPD样本中的表达与对照组相比显著增加。
这些发现揭示了COPD患者中特定mRNAs的表达变化,为理解COPD的分子病理提供了新的视角。IL-6的增加可能与COPD中的炎症反应有关,而CAT的降低可能与氧化应激的增加相关;TEP1的增加可能与COPD中的端粒酶活性改变有关,这可能影响细胞的老化和凋亡;SMAD7的降低可能与TGF-β信号通路的失调有关,这是COPD中已知的关键病理过程[14]。
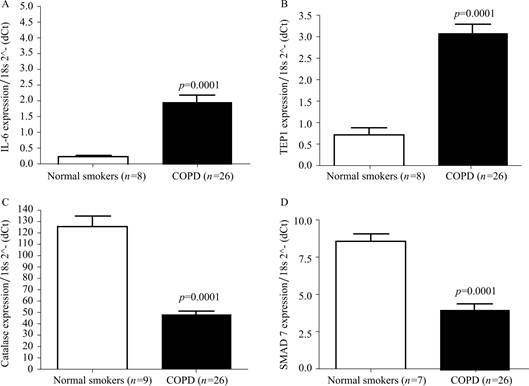
图2 qRT-PCR验证差异表达的mRNAs[1]
Fig.2 qRT-PCR verification of differentially expressed mRNAs[1]
结合上述实验分析,进一步采用TGFβ处理和迁移分析方法,对COPD患者肺部的miRNAs和mRNAs进行了深入研究。通过这些方法,得到了与无COPD的吸烟者相比,COPD受试者肺中Top10上调和下调的miRNAs和mRNAs的结果,这些结果分别展示在表2、表3、表4和表5中。它们揭示了在COPD组织和吸烟者组织之间表达差异最大的10个基因,并提供了其在没有气流限制的情况下已知的生物学功能。例如,白细胞介素6(IL-6)在COPD样本中的表达量增加,而过氧化氢酶(CAT)的表达量减少;端粒酶相关蛋白1(TEP1)在COPD样本中的表达显著增加,这可能与端粒酶活性的改变有关;SMAD7在COPD的发病机制中已被激活,根据微阵列分析,其表达显著降低,并通过qRT-PCR验证。
表2 COPD受试者肺中Top10上调的microRNAs与无COPD的吸烟者比较[1]
Table 2 Comparison of Top10-upregulated microRNAs in the lungs of COPD subjects and smokers without COPD[1]
|
miRNA |
Fold difference(COPD/normal smokers) |
p Value |
pFDR value |
|
hsa-miR-223 |
2.93 |
6.63E-04 |
1.37E-03 |
|
hsa-miR-1274a |
2.73 |
3.19E-10 |
4.44E-09 |
|
hsa-miR-144 |
2.38 |
2.83E-03 |
4.26E-03 |
|
hsa-miR-374a |
2.25 |
1.72E-04 |
4.74E-04 |
|
hsa-miR-664 |
2.21 |
2.00E-08 |
2.02E-07 |
|
hsa-miR-148a |
2.15 |
2.34E-07 |
1.68E-06 |
|
hsa-miR-766 |
2.06 |
1.05E-05 |
4.76E-05 |
|
hsa-miR-486-5p |
2.05 |
3.26E-04 |
8.21E-04 |
|
hsa-miR-10a |
2.05 |
7.47E-04 |
1.46E-03 |
|
hsa-miR-451 |
2.00 |
5.39E-02 |
4.59E-02 |
表3 COPD受试者肺中Top10下调的microRNAs与无COPD的吸烟者比较[1]
Table 3 Comparison of top 10 down-regulated microRNAs in the lungs of COPD subjects and smokers without COPD[1]
|
miRNA |
Fold difference(COPD/normal smokers) |
p Value |
pFDR value |
|
hsa-miR-923 |
-5.06 |
5.88E-06 |
2.91E-05 |
|
hsa-miR-937 |
-2.36 |
1.89E-06 |
1.08E-05 |
|
hsa-miR-422a |
-2.12 |
1.45E-06 |
8.74E-06 |
|
hsa-miR-576-3p |
-2.06 |
1.57E-06 |
9.22E-06 |
|
hsa-miR-513a-5p |
-2.06 |
3.57E-04 |
8.54E-04 |
|
hsa-miR-25* |
-2.01 |
2.10E-07 |
1.56E-06 |
|
hsa-miR-99b* |
-1.81 |
1.23E-05 |
5.25E-05 |
|
hsa-miR-125b-1* |
-1.75 |
1.38E-05 |
5.70E-05 |
|
hsa-miR-24 |
-1.75 |
1.59E-04 |
4.49E-04 |
|
hsa-miR-187* |
-1.64 |
3.14E-04 |
8.04E-04 |
表4 与无COPD的吸烟者相比,患有COPD的受试者的肺组织中前10种上调的mRNA[1]
Table 4 Top 10 upregulated mRNAs in lung tissue of subjects with COPD compared to smokers without COPD[1]
|
Gene ID |
Gene title |
Gene symbol |
Fold difference (COPD/normal Smokers) |
p Value |
pFDR value |
Function |
|
9507 |
ADAM metallopeptidase with thrombospondin type 1 motif,4 |
ADAMTTS4 |
8.91 |
2.16E-04 |
4.02E-04 |
Proteolytic activity |
|
7011 |
Telomerase-associated Protein 1 |
TEP1 |
7.24 |
4.88E-13 |
5.06E-11 |
Alternative splicing,nucleo-cytoplasmic transport |
|
10949 |
Heterogeneous nuclear ribonucleoprotein A0 |
HNRNPAO |
6.86 |
8.02E-22 |
5.24E-18 |
mRNA processing transport and metabolism |
|
2304 |
Forkhead box E1(thyroid transcription factor 2) |
FOXE1 |
6.30 |
4.02E-05 |
1.01E-04 |
Transcription factor,thyroid gland organogenesis |
|
4001 |
Lamin B1 |
LMNB1 |
6.05 |
8.44E-12 |
4.52E-10 |
Nuclear stability chromatin structye |
|
117245 |
HRAS-like suppressor family,member 5 |
HRASLS5 |
5.26 |
1.59E-17 |
1.44E-14 |
Transferase,catalytic activity |
|
3596 |
Interleukin 6(interferon,beta 2) |
IL6 |
5.18 |
1.50E-03 |
2.01E-03 |
Inflammation,B cell Differentiation |
|
6280 |
S100 calcium binding Protein A9 |
S100A9 |
5.14 |
8.17E-07 |
4.14E-06 |
Cell cycle peogression,differentiation,immune system |
|
6696 |
Secreted phosphoprotein 1(osteopontin,bone Sialoprotein I,early T lymphocyte activation 1) |
SPP1 |
5.10 |
6.58E-05 |
1.51E-04 |
Metabolic process,cellular organization,binding |
|
27097 |
TAF5-like RNA polymerase Ⅱ,p300/CBP-associated factor(PCAF)-associated factor,65 kDa |
TAF5L |
4.95 |
2.05E-12 |
1.51E-10 |
Histone acetylase |
表5 与无COPD的吸烟者相比,患有COPD的受试者的肺组织中前10种下调的mRNA[1]
Table 5 Top 10 down-regulated mRNAs in lung tissue of subjects with COPD compared to smokers without COPD[1]
|
Gene ID |
Gene title |
Gene symbol |
Fold difference (COPD/normal smokers) |
p Value |
pFDR value |
Function |
|
22870 |
SAPS domain family,member 1 |
SAPS1 |
-4.46 |
3.15E-03 |
3.69E-03 |
Protein phosphatase |
|
6435 |
Surfactant,pulmonary-associated protein A1B |
SFTPA1B |
-4.23 |
2.90E-08 |
2.76E-07 |
Binds surfactant phospholipids |
|
5507 |
Protein phosphatase 1,regulatory(inhibitor)subunit 3C |
PPP1R3C |
-4.17 |
3.99E-05 |
1.01E-04 |
Glycogen-targeting subunit,increases glycogen |
|
2662 |
Growth differentiation factor 10 |
GDF10 |
-4.14 |
3.07E-05 |
8.04E-05 |
TGFβ signalling |
|
5350 |
Phospholamban |
PLN |
-3.93 |
1.78E-06 |
7.72E-06 |
Calcium regulation |
|
25928 |
Sclerostin domain containing 1 |
SOSTDC1 |
-3.87 |
2.68E-04 |
4.79E-04 |
Inhibits TGFβ signaling and enhances Wnt siqnalling |
|
9353 |
Slit homologue 2 (Drosophila) |
SLIT2 |
-3.48 |
4.32E-08 |
3.81E-07 |
Cell migration and axonal navigation |
|
4094 |
v-maf musculoaponeurotic fibrosarcoma oncogene homoloque(avian) |
MAF |
-3.46 |
1.41E-06 |
6.35E-06 |
Transcriptional activator or repressor,oncogene or tumor suppressor depending on context |
|
123624 |
ATP/GTP binding protein-like 1 |
AGBL1 |
-3.37 |
7.95E-06 |
2.66E-05 |
Tubulin processing |
|
4629 |
Myosin,heavy chain 11,smooth muscle |
MYH11 |
-3.31 |
2.09E-06 |
8.78E-06 |
Contractile protein |
通过miRNA和mRNA表达谱富集分析,继续深入探讨latent TGF-β和Wnt通路在COPD中的作用。利用TargetScan、miRanda和PicTar这三个预测工具,确定了假定的miRNA相互作用靶点,以揭示差异表达的miRNA(DEmiRNA)和差异表达基因(DEGs)之间的相互作用。通过序列互补匹配和保守性分析,预测miRNA与其靶基因之间的潜在联系。研究结果表明,TGF-β和Wnt通路在COPD的发病机制中确实发挥作用[15,16]。例如,Wnt信号在肺发育、再生和疾病进展中起着重要作用,特别是在肺纤维化中,Wnt信号与TGF-β信号通路之间存在交叉作用。在COPD中,TGF-β1能够激活WNT/β-catenin信号,增加肺成纤维细胞中ECM的产生,并促进肌成纤维细胞分化。此外,WNT/β-catenin信号通路的激活与COPD中的异常ECM转换有关。
已知miR-15b能够靶向支气管上皮细胞中的SMAD7,进而影响TGF-β信号传导。研究发现,在COPD患者与无COPD的吸烟者肺组织中,有70个miRNA和2667个mRNA表现出差异表达。通过miRNA和mRNA表达谱富集分析,研究揭示了与COPD发病机制可能相关的生物学通路,包括转化生长因子β(TGF-β)、Wnt和黏着斑通路。特别地,与无气流阻塞的吸烟者相比,miR-223和miR-1274a在COPD受试者中受到显著影响,COPD样本中miR-15b的表达升高,且这种升高局限于肺气肿和纤维化两个区域。在COPD的GOLD类别中,miR-15b的差异表达也得到了观察,SMAD7被验证为miR-15b的靶标,在COPD的支气管上皮细胞中表达降低。
通过对疾病网络(或疾病体)和相互作用网络(由蛋白质相互作用网络定义)进行数据挖掘,研究明确了COPD的调控网络,如图3所示。这些发现不仅加深了对COPD中miRNA和mRNA相互作用的理解,而且为未来治疗策略的开发提供了潜在的分子靶点。
研究在慢性阻塞性肺疾病(COPD)中,观察到一系列基因表达的变化,这些变化涉及关键的信号通路和生物过程。具体来说,血栓素1(THBS1)、抑制素βB(INHBB)、Rho相关螺旋-环化蛋白激酶
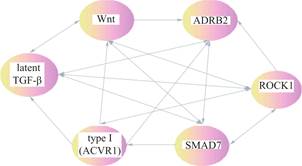
图3 COPD的调控网络
Fig.3 Network regulation of COPD
2(ROCK2)、SMAD特异性E3泛素蛋白连接酶1(SMURF1)、骨形态发生蛋白7(BMP7)和E2F转录因子4(E2F4)在COPD中的表达上调(用红色表示),而激活素A受体1型(ACVR1)、SMAD7、ROCK1、潜伏TGF-β结合蛋白1(LTBP1)、TGF-β受体1(TGFBR1)和TGFBR2的表达下调(用绿色表示)。
这些基因的表达变化反映了COPD病理过程中的重要分子机制,THBS1和INHBB在细胞外基质形成和组织修复中起作用;ROCK2和SMURF1则与细胞信号传导和蛋白质降解相关;BMP7和E2F4在细胞分化和增殖中扮演关键角色;相反,ACVR1、SMAD7、ROCK1、LTBP1、TGFBR1和TGFBR2的下调可能与TGF-β信号通路的抑制有关,这是COPD中纤维化和炎症反应的关键调控因素。此外,在同一组织中,Wnt和粘附斑块通路的几个差异表达基因被发现是差异表达miRNA的潜在靶点,这进一步揭示了COPD中miRNA调控网络的复杂性,如图4所示[1]。
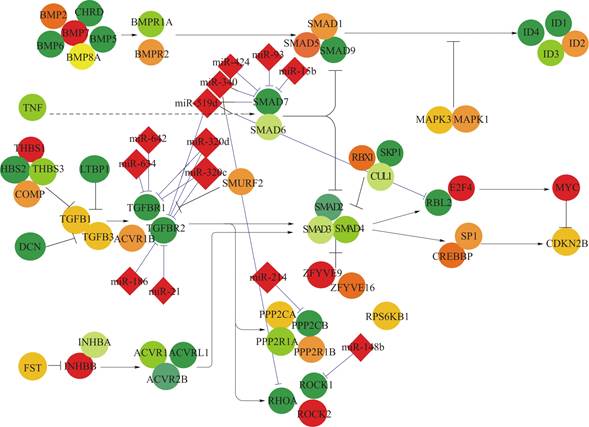
图4 COPD的发病机制[1]
Fig.4 Pathogenesis of COPD[1]
3 敏感基因识别
临床专家已经识别出16种与COPD经常相关的常见疾病[6],这些疾病之间的联系是基于共享基因或蛋白质的改变,以及这些改变的蛋白质是否在相互作用组中相互连接,如表6。这种连接性构成了疾病之间的网络,被称为“双体”,它代表了疾病间的关联性。从研究进一步这些疾病中筛选出与COPD形成“双体”关系的疾病体,更深入地理解COPD的复杂病理机制,并揭示新的可能治疗靶点。通过网络分析,识别出与COPD病理生理过程密切相关的其他疾病,从而为COPD的综合管理提供更全面的视角。
表6 十九种经常与COPD相关的常见疾病
Table 6 Nineteen common diseases frequently associated with COPD
|
疾病类型 |
疾病 |
CUIs(CUI) |
基因 |
|
慢性阻塞性肺病 |
|||
|
呼吸道疾病 |
慢性支气管炎 |
16 |
11 |
|
|
慢性阻塞性肺病 |
14 |
181 |
|
|
肺气肿 |
33 |
11 |
|
慢性阻塞性肺病相关疾病 |
|||
|
营养和代谢疾病 |
糖尿病 |
332 |
757 |
|
|
代谢综合征 |
4 |
47 |
|
|
肥胖 |
47 |
313 |
|
肌肉骨骼疾病 |
肌肉无力 |
8 |
65 |
|
|
骨质疏松 |
37 |
128 |
|
呼吸道疾病 |
肺动脉高压 |
22 |
57 |
|
|
肺癌 |
81 |
1369 |
|
|
睡眠呼吸暂停 |
19 |
27 |
|
偏侧和淋巴疾病 |
贫血 |
5 |
13 |
|
|
红细胞增多症 |
11 |
16 |
|
心血管疾病 |
心房颤动 |
21 |
74 |
|
|
心力衰竭 |
73 |
410 |
|
|
缺血性心脏病 |
151 |
618 |
|
|
中风 |
57 |
189 |
|
病理状况、体征和症状 |
恶病质 |
8 |
19 |
|
行为和行为机制 |
抑郁症 |
105 |
160 |
通过Pathway Commons数据库[6],研究收集了与COPD相关的基因数据,并据此建立了一个diseasome网络。在该网络中,如果两种疾病共享基因或蛋白质的改变,或者这些改变的蛋白质在相互作用组中相互连接,则称这两种疾病是相连的,即它们构成了所谓的“双体”。例如,在面板A中,可以看到四种不同的疾病(D1、D2、D3和D4)共享相关基因(GA、GB、GC)。利用这些信息,可以构建一个diseasome,其中D1与D2共享基因GA,D3与D4共享基因GC。进一步地,在面板B中,将上述遗传信息与相互作用组相结合,以构建双体体。例如,由基因GB(编码蛋白质PB)与由基因GC(编码蛋白质PC)的蛋白质相互作用,这种相互作用在diseasome中得到了体现。
继续讨论两种疾病之间的共同蛋白质双体的构建情况。定义分子合并症指数(MCI),目的在于估计两种给定疾病在分离体中的关联强度时最小化中间的偏差,计算公式如下:
MCIdis1,dis2=![]()
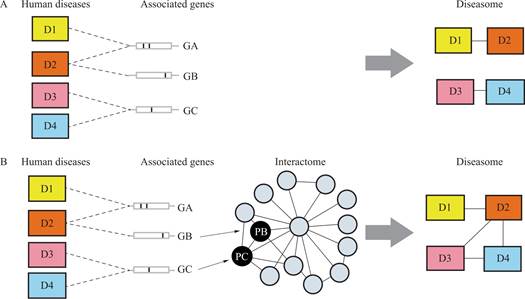
图5 两种不同疾病的共享基因[2]
Fig.5 Genes shared by two different diseases[2]
在得到共享基因和共享蛋白之后,需要进行多病蛋白的功能分析,以确定两种疾病之间共享蛋白质最重要的生物学功能。调用Reactome的生物学分析[7],具体为R包Reacto-mePA,它使用超几何函数来测试注释的重要性[11]。同时使用Jaccard系数评估慢性阻塞性肺病多病症共有的生物学通路数量,目的是衡量两种COPD多种疾病(例如肺癌-COPD和糖尿病-COPD)在生物学途径水平上的相似程度的指标。根据 Grosdidier S等人发现,在成对的慢性阻塞性肺病和几种心血管疾病(包括心力衰竭、缺血性心脏病和中风)中,Jaccard系数值最高,它们共享约50%的受累通路,如图6所示[2]。同样,成对的CVD-COPD和成对的COPD伴有营养和代谢疾病之间的通路重合水平也相当大。令人惊讶的是,研究观察到CVD-COPD对与骨质疏松症-COPD之间存在相对较高的Jaccard系数,以及肺癌-COPD和糖尿病-COPD的通路相似性为48%(肺癌和糖尿病不考虑COPD的通路相似性仅为35%)。尽管程度较轻,但营养和代谢疾病(COPD)和肌肉骨骼疾病(COPD)组也呈现出通路相似性。从这项分析中可以得出一些有趣关联,包括抑郁症-慢性阻塞性肺病和糖尿病-慢性阻塞性肺病之间的重叠,以及抑郁症-慢性阻塞性肺病和心力衰竭-慢性阻塞性肺病之间的重叠。同样令人感兴趣的是,一些共病-COPD对涉及的生物学途径与其他疾病-COPD对的生物学途径几乎没有关系(例如,贫血-COPD、恶病质-COPD、睡眠-呼吸暂停-COPD或红细胞增多症-COPD)如图6所示,考虑生物学途径,每个细胞都根据 Jaccard系数的值进行着色,该系数表示两种COPD多种疾病之间的相似程度。这些观察结果突出了慢性阻塞性肺病多发病在生物途径水平上的有趣关联。
Jaccardcoefficientmultimorbidity1,multimorbidity2
=![]()
得到COPD通过基因和蛋白质与其合并症有关这一结论后,在疾病的遗传基础上,Yoshida T等人结合从相互作用组获得的数据构建COPD二分体[5]。发现研究中的16种合并症都可以通过在细胞网络内相互作用的共享基因和蛋白质来连接,得到分子水平上不同疾病体的联系,如图7所示。该网络表示疾病和蛋白质之间的关联(薄边)以及蛋白质之间的关联(厚边)。用绿色显示与慢性阻塞性肺病和贫血直接相关的蛋白质(SOD2、TP53和TNF)。粉红色淋巴结和里拉淋巴结分别是与慢性阻塞性肺病或贫血相关的蛋白质,但因为这些蛋白质在相互作用组内相互作用,故也可以作为两种疾病之间的桥梁。
然而,在COPD双体中,共享基因和蛋白质的数量范围从不到100个到超过700。对此,引出分子合并症指数MCI,它是判断两种疾病之间关联强度的指标。对于将研究较好的疾病(肺癌等)进行标准化后发现,缺血性心脏病在COPD多病列表中排名第一(大约400种共享蛋白质,MCI为0.53),其次是肺癌,中风和糖尿病。MCI的频率分布如图8所示。
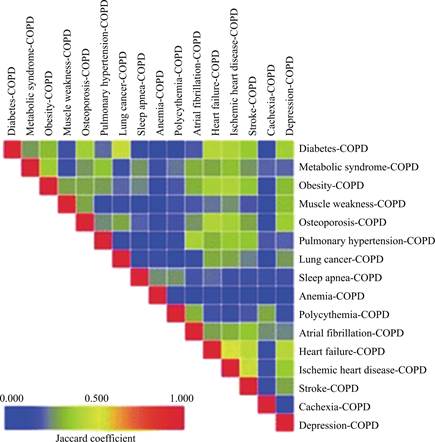
图6 慢性阻塞性肺病多病症之间的相似性[2]
Fig.6 The similarities between multiple conditions of chronic obstructive pulmonary disease[2]
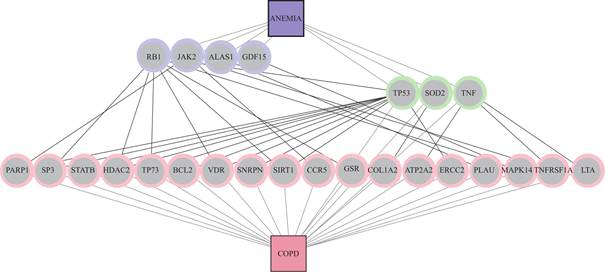
图7 慢性阻塞性肺病和贫血在分子水平上的联系[2]
Fig.7 The relationship between chronic obstructive pulmonary disease and anemia at the molecular level[2]
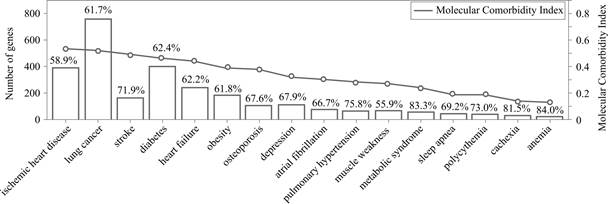
图8 慢性阻塞性肺病与此处研究的每种多种疾病之间共享的蛋白质数量(条)及其相应的MCI(线)[2]
Fig.8 Number of proteins(strips)and their corresponding MCI(lines)shared between COPD and each of the multiple diseases studied here[2]
Plus特定基因及其与COPD及多病症疾病关联:ADRB2。
![]() 有助于胰腺分泌胰岛素。
有助于胰腺分泌胰岛素。
![]() 影响患者对支气管扩张剂治疗的反应。
影响患者对支气管扩张剂治疗的反应。
![]() 在血管中,它扩张冠状动脉和骨骼肌动脉。
在血管中,它扩张冠状动脉和骨骼肌动脉。
![]() 在呼吸方面,参与支气管中的平滑肌松弛,从而促进呼吸。
在呼吸方面,参与支气管中的平滑肌松弛,从而促进呼吸。
对ADRB2多态性的总结:Drier Y等人发现COPD伴心血管疾病(心房颤动、心力衰竭、缺血性心脏病和中风),以及糖尿病、肺癌和肥胖症。在特定的人群中:希腊人群的R16G多态性与FEV1(1秒用力呼气容积)的COPD严重程度之间存在显著相关性[10]。有趣的是,R16G-Q27E单倍型已被证明与肥胖、绝经后妇女的葡萄糖耐量和胰岛素敏感性有关[8];丹麦人群的ADRB2 T164I多态性与一般人群肺功能下降和COPD风险增加有关[13]。对于特定患者出现这些并发症,使用网络医学的新学科进行分析,系统地探索一种特定疾病的分子复杂性,具有识别明显疾病之间新分子关系的潜力不同[3]。在一般人群中,这种多态性还与血压升高、高血压发生率增加和缺血性心脏病风险增加有关。ADRB2 基因与多种疾病相关:慢性阻塞性肺病伴心血管疾病(心房颤动、心力衰竭、缺血性心脏病和中风),以及糖尿病、肺癌和肥胖症。同样令人感兴趣的是,由该基因编码的蛋白质已被证明参与支气管平滑肌松弛,从而促进呼吸[12];在血管中,它扩张冠状动脉和骨骼肌动脉[8]并且它也有助于胰腺分泌胰岛素[9]。ADRB2 一直是 COPD 药物基因组学研究的焦点:该基因的几种多态性已被证明影响患者对支气管扩张剂治疗的反应[10]。因此,ADRB2 T164I多态性可以解释COPD与心血管疾病之间的关联。总而言之,ADRB2基因代表了一个有趣的候选基因,可以解释COPD与糖尿病、肥胖症和缺血性心脏病的多种疾病。
上述结果表明,16种流行的COPD多病症通常与慢性阻塞性肺病共享基因、蛋白质和生物学途径,最普遍的慢性阻塞性肺病多病症在分子水平上是相关的。Thorn D等人的研究中提到上述中许多蛋白质是烟草烟雾化学物质的靶标[4]。而这些观察结果支持了COPD双体的共享成分假说,并允许识别COPD与其合并症之间的特定分子联系(基因、蛋白质、生物途径)。COPD的多种疾病可能应该被理解为一个临床表现网络(即差异体),它表达了许多环境、生物学和遗传(及表观遗传学)控制因素之间的复杂相互作用。而烟草烟雾是COPD及其一些相关疾病的主要危险因素。研究表明,与COPD有关的基因有六个,分别为latent TGF-β,Wnt,typeI(ACVR1),ADRB2,SMAD7,ROCK1。其中按敏感度从高到低排序,latent TGF-β最敏感,为敏感基因。
4 功能分析与预警
在慢性阻塞性肺疾病(COPD)的病理过程中,Wnt、latent TGF-β、type I(ACVR1)、SMAD7、ROCK1、ADRB2等关键生物标志物扮演着重要角色。这些指标之间的相互作用和调控机制,为研究提供了深入理解COPD病理生理的机会。通过系统性地研究这些“共享组件”,可以揭示它们在COPD多发病中的潜在作用,这对于早期诊断、预后评估和治疗策略的优化具有重要意义。
具体来说,Wnt信号通路与TGF-β家族成员在纤维化过程中存在交叉调控,Wnt信号的激活对于TGF-β介导的纤维化是必需的。此外,ACVR1作为TGF-β家族的一种type I受体,在信号传导中起着核心作用,其突变可能导致疾病的发展。SMAD7作为抑制性SMAD,可以与YAP和β-catenin相互作用,影响TGF-β和Wnt信号通路。ROCK1和ADRB2则可能通过影响细胞内信号传导和功能,参与COPD的病理过程。
(1)Wnt信号通路在COPD的发病机制中占据核心地位。值得注意的是,Wnt/β-连环蛋白信号通路的异常激活与COPD及特发性肺纤维化等慢性肺部疾病的发生发展有着密切的联系。因此,针对这一信号通路的靶向治疗策略有望为COPD的预防和治疗提供创新的解决方案。
(2)Latent TGF-β(转化生长因子-β)在COPD中扮演着至关重要的角色:作为细胞因子的关键成员,TGF-β在COPD的病理生理过程中起着核心作用。在COPD患者中,TGF-β的表达和激活水平升高,与肺部炎症、组织重塑(包括纤维化和肺气肿)有着直接的联系。TGF-β信号通路的异常激活不仅参与了COPD的进展,而且可能成为治疗的新靶点。深入研究TGF-β信号通路有助于深化本研究对COPD发病机制的理解,并可能为开发新疗法提供新思路。
(3)ACVR1,编码TGF-β受体超家族中的骨形态发生蛋白(BMP)类型I受体,可能通过调节TGF-β信号,对COPD的病程产生影响。
(4)SMAD7是TGF-β/SMAD信号通路中的一个重要负反馈调节因子,已知其在心脏纤维化中具有抑制作用。在COPD患者肺部,SMAD7的表达水平变化,这表明SMAD7/TGF-β通路可能在COPD的发展中扮演着关键角色。
(5)ROCK1是一种丝氨酸/苏氨酸蛋白激酶,参与多种细胞过程,包括细胞骨架重组、细胞增殖和迁移等。在COPD的背景下,ROCK1可能参与调节肺部炎症和纤维化的过程。
(6)ADRB2基因是一个引人注目的候选基因,可能解释COPD与糖尿病、肥胖症和缺血性心脏病等多种疾病的关联。ADRB2基因的某些变异可能与COPD的发病风险和治疗反应相关。
在对预警指标进行深入理解后,结合Ezzie ME和 Grosdidier S等人的研究,可以将敏感基因latent TGF-β(转化生长因子-β)视为COPD预警系统中的核心指标,而其他指标则作为辅助指标。通过设定特定的阈值和干预方案,主要干预措施包括调整药物治疗方案、加强氧疗以及开展肺康复训练等[1,2]。结合医学网络分析和调控模型,以实时监控latent TGF-β等六个关键预警指标的动态变化,同时还需监督外部体征的变化,如吸烟情况、肺功能测试结果、紫绀现象、呼吸急促、心率加快等,以及有无下肢水肿、动脉血气分析结果、心理状况和体重变化等。
在这一监测体系中,研究将阈值设定为0.7886(fold change数)。如果患者的latent TGF-β基因表达与正常人相比fold change数超过这一阈值,则视为超出预警,提示病情可能加重;如果未超过,则认为未超出预警。通过这种综合监测和管理策略,能够更精确地控制COPD的进展,优化患者的治疗和生活质量。
5 结束语
本文聚焦于慢性阻塞性肺疾病(COPD)相关的六个敏感基因,基于先前研究的成果构建数据挖掘和网络调控模型,复现了参考文献中的关键发现,并对所得结论进行了进一步的验证和优化。研究结果确认了COPD调控网络中的核心基因,包括latent TGF-β、Wnt、typeI(ACVR1)、ADRB2、SMAD7和ROCK1。在这些基因中,latent TGF-β因其高度的敏感性而被识别为最关键的敏感基因。
通过方法论的深化和模型的精细化,本文强化了对COPD调控网络的理解,同时为未来针对这些关键基因的靶向治疗提供了坚实的理论基础。通过分析与疾病相关的社会行为和生活方式数据,识别与之相关的RNA或其他生物标志物,利用网络调控和追踪技术,构建疾病的调控网络。进一步地对基因表达数据进行分析,对敏感基因进行敏感度排序,设定基因表达的阈值和相应的干预方案,实现对疾病的预警分析。这种综合分析框架为COPD的生物标志物发现、疾病机制解析以及新疗法的开发开辟了新的道路,且对于其他具有复杂调控网络和多因素影响的疾病研究也具有指导意义。面对现实因素之间错综复杂的联系,可以通过简化模型来提高计算效率,同时引入更多的变量和相互作用来增强模型的准确性,从而得出更具普遍性的结论。这种平衡模型复杂性和计算效率的方法,将有助于在保持模型准确性的同时提高其在实际应用中的可行性和实用性,更有效地理解和预测疾病的发展,为患者提供更精准的医疗干预。
利益冲突: 作者声明无利益冲突。
[①] *通讯作者 Corresponding author:王浩华,huazi8112@hainanu.edu.cn
收稿日期:2024-12-12; 录用日期:2025-01-12; 发表日期:2025-03-28
基金项目:国家自然科学基金(12261028);海南省自然科学基金(122QN215);海南省工程建模与统计计算重点实验开放课题(HNGCTJ2401)。
参考文献(References)
[1] Ezzie ME, Crawford M, Cho JH, et al. Gene expression networks in COPD: microRNA and mRNA regulation [J]. Thorax, 2012, 67(2): 122-131.
https://doi.org/10.1136/thoraxjnl-2011-200089.
[2] Grosdidier S, Ferrer A, Faner R, et al. Network medicine analysis of COPD multimorbidities[J]. Respir Res, 2014, 15(1): 111.
https://doi.org/10.1186/s12931-014-0111-4.
[3] Drier Y, Sheffer M, Domany E. Pathway-based personalized analysis of cancer[J]. Proc NatlAcad Sci, 2013, 110(16): 6388-6393.
https://doi.org/10.1073/pnas.1219651110.
[4] Dm M, Thorn D, Swensen A, et al. Prevalence and outcomes of diabetes, hypertension and cardiovascular disease in COPD[J]. Eur Respir J, 2008, 32: 962-969.
https://doi.org/10.1183/09031936.00012408.
[5] Yoshida T, Tuder R M. Pathobiology of cigarette smokeinduced chronic obstructive pulmonary disease[J]. Physiol Rev, 2007, 87: 1047-1082.
https://doi.org/10.1152/physrev.00048.2006.
[6] Bauer-Mehren A, Furlong LI, Sanz F. Pathway databases and tools for their exploitation: benefits, current limitations and challenges[J]. Mol Syst Biol, 2009, 5: 290.
https://doi.org/10.1038/msb.2009.47.
[7] Croft D, OKelly G, Wu G, et al. Reactome: Reaction, pathways, and biological process databases[J]. Nucleic acid research, 2011, 39: D691-D697.
https://doi.org/10.1093/nar/gkq1018.
[8] Prior S J, Goldberg A P, Ryan A S. ADRB2 haplotype is associated with glucose tolerance and insulin sensitivity in obese postmenopausal women[J]. Obesity(Silver Spring), 2011, 19: 396-401.
https://doi.org/10.1038/oby.2010.182.
[9] Lacey R J, Cable H C, James RF, et al. Concentration-dependent effects of adrenaline on the profile of insulin secretion from isolated human islets of Langerhans[J]. J Endocrinol 1993, 138: 555-563.
https://doi.org/10.1677/joe.0.1380555.
[10] Hersh C P. Pharmacogenetics of chronic obstructive pulmonary disease: challenges and opportunities[J]. Pharmacogenomics, 2010, 11: 237-247.
https://doi.org/10.2217/pgs.09.176.
[11] Yu G, Reactome P A. Reactome Pathway Analysis R package version 2. 15. 2[Z]. 2012.
https://doi.org/10.18129/B9.bioc.ReactomePA.
[12] Thomsen M, Dahl M, Tybjaerg-Hansen A, et al. β2-adrenergic receptor Thr164IIe polymorphism, blood pressure and ischaemic heart. disease in 66 750 individuals [J]. J Intern Med, 2012, 271: 305-314.
https://doi.org/10.1111/j.1365-2796.2011.02451.x.
[13] Thomsen M, Nordestgaard B G, Sethi A A, et al. 2-adrenergic receptor polymorphisms, asthma and COPD: two large population-based studies[J]. EurRespir J, 2012, 39: 558-566.
https://doi.org/10.1183/09031936.00163611.
[14] Zandvoort A, Postma D S, Jonker M R, et al. Altered expression of the Smad signalling pathway: implications for COPD pathogenesis[J]. Eur Respir J, 2006; 28: 533e41.
https://doi.org/10.1183/09031936.06.00104905.
[15] Barnes P J. The cytokine network in chronic obstructive pulmonary disease[J]. Am J Respir Cell Mol Biol, 2009, 41: 631e8.
https://doi.org/10.1165/rcmb.2009-0201TR.
[16] Kneidinger N, Yildirim A O, Callegari J, et al. Activation of the WNT/beta-catenin pathway attenuates experimental emphysema[J]. Am J Respir Crit Care Med, 2011, 183: 723e33.
https://doi.org/10.1164/rccm.201003-0424OC.
Research on Chronic Obstructive Pulmonary Disease from the Perspective of Network Regulation
(1. School of Mathematics and Statistics, Hainan University, Haikou 570228, China
2. School of International Tourism and Public Management, Hainan University, Haikou 570228, China
3. Key Laboratory of Engineering Modeling and Statistical Computing of Hainan Province, Hainan University, Haikou 570228, China)
Abstract: Chronic obstructive pulmonary disease (COPD) is a widespread and profoundly impactful respiratory illness, posing a significant threat to public health. Unraveling its complex pathogenesis and the intricate regulatory networks it entails is essential. This comprehensive article sets forth to meticulously delineate the regulatory network of COPD, engaging in a thorough and rigorous examination of its multifaceted ties to various biological pathways. Anchored in a rich tapestry of clinical data and bolstered by a broad synthesis of leading-edge scientific literature, the study employs cutting-edge data mining techniques and sophisticated network regulatory models to construct a protein-protein interaction (PPI) network. In this process, fully utilizing the data information covered by smoking exposure, accurately identifying the associated RNA and miRNA, and laying a solid foundation for subsequent research. By integrating state-of-theart network regulation and tracking technologies, the researchers have meticulously crafted a COPD regulatory network with latent TGF-β, Wnt, typeI (ACVR1) , ADRB2, SMAD7, and ROCK1 as its linchpin nodes. Subsequently, high-precision gene expression data analysis was used to evaluate the sensitivity of these six genes closely related to COPD, and their sensitivity was ranked from high to low according to strict criteria. The results showed that late TGF-β had the highest sensitivity and was confirmed as a sensitive gene. In summation, by establishing scientifically sound gene expression thresholds and crafting intervention strategies that align with these benchmarks, the study achieves a formidable early warning analysis for COPD. This analysis is pivotal for the diseases early diagnosis, a critical juncture in enhancing patient prognoses. Furthermore, it lays a formidable scientific foundation for the development of precision treatments, which could herald a new era in the clinical management of COPD. It is anticipated that these insights will not only deepen our comprehension of COPD but also drive forward clinical practices, potentially elevating the standard of care for those afflicted with this debilitating disease to unprecedented heights.
Keywords: COPD, sensitive genes, early warning analysis, network regulation, data mining
DOI: 10.48014/jcss.20241212002
Citation: CHEN Qiuyu, LI Xinlei, WANG Huaixing, et al. Research on chronic obstructive pulmonary disease from the perspective of network regulation[J]. Journal of Chinese Statistical Sciences, 2025, 3(1): 1-14.